È online il quattordicesimo contributo dedicato alla promozione dei risultati della ricerca dei nostri dottorandi.
Anna Schroeder - Corso di Dottorato in Ambiente e vita
DNA metabarcoding vs. analisi morfologica – Analisi dello zooplancton in acque di transizione
La analisi della biodiversità dello zooplancton è un elemento cruciale nel monitoraggio degli ecosistemi marini e della risposta delle comunità alle alterazioni ambientali. Il DNA metabarcoding tramite sequenziamento di seconda generazione sta emergendo come un importante strumento per il monitoraggio della biodiversità (Taberlet et al., 2012). Con tale tecnica è infatti possibile identificare una vasta gamma di taxa in tempi molto più rapidi rispetto al classico approccio morfologico, permettendo quindi di incrementare lo sforzo di campionamento (frequenza temporale e copertura spaziale) a costi sostenibili (Brannock et al., 2014; Coissac et al., 2012).
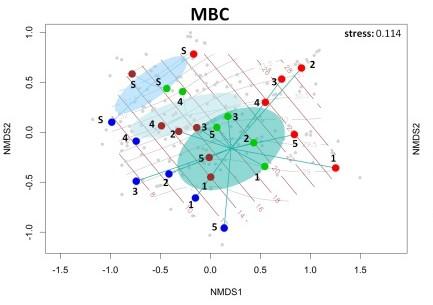
L’obiettivo di questo studio è di valutare l’efficacia del DNA metabarcoding per la stima della biodiversità dello zooplancton nelle acque di transizione (lagune, estuari, ecc.). A tale scopo sono stati condotti campionamenti stagionali di zooplancton in Laguna di Venezia e nella zona costiera antistante (Nord Adriatico). Ciascun campione è stato diviso in due parti omogenee, per la successiva determinazione sia con stereomicroscopio (approccio morfologico) sia tramite DNA metabarcoding utilizzando un frammento (313 bp) della citocromo c ossidasi subunità I (Leray et al., 2013). L’analisi molecolare si è rivelata molto efficacie nel descrivere la complessità della comunità zooplanctonica, permettendo di identificare un maggior numero di taxa rispetto al metodo morfologico (224 vs 88 taxa). Questa differenza è riconducibile in particolare alla capacità dell’approccio molecolare di identificare le specie criptiche e discriminare il meroplancton, identificato con il metodo morfologico solo a livello di ordine. Nonostante queste differenze, a livello ecologico i due metodi hanno fornito indicazioni comparabili. I risultati dell’analisi della biodiversità, misurata tramite l’indice di Shannon-Wiener, hanno evidenziato una correlazione significativa tra metabarcoding e identificazione morfologica. Inoltre, I due metodi hanno evidenziato i medesimi pattern spazio-temporali, riconducibili principalmente alla stagionalità, seguendo gradienti di temperatura, e alla localizzazione delle stazioni, seguendo il gradiente di salinità dal mare verso le zone più interne e influenzate dalle acque dolci, tipico delle acque di transizione (Fig. 1).
Nel confrontare i risultati dell’analisi della biodiversità ottenuti con i due metodi, va tenuto in considerazione che con l’approccio molecolare si utilizza il numero di sequenze come proxy della biomassa (Lindeque et al., 2013), mentre l’approccio morfologico si basa sulla stima dell’abbondanza, ovvero sul conteggio del numero di individui. La ricerca di una correlazione tra numero di sequenze e abbondanza (o biomassa) è stata oggetto di numerosi studi, in generale ottenendo correlazioni molto deboli (es. Harvey et al., 2017). Diversamente, in linea con i risultati ottenuti da Bucklin et al. (2019) per il marker 18S (V9), in questo studio sono emersi risultati promettenti anche per il COI marker. Il numero di sequenze è infatti risultato significativamente correlato all’abbondanza per le specie identificate con entrambi i metodi, per le classi più abbondanti di artropodi e per la maggior parte dei phyla (Fig. 2).
I risultati di questo studio indicano quindi come il DNA metabarcoding sia un efficiente strumento per la stima della biodiversità, in particolare negli studi condotti in ambienti lagunari ed estuarini, dove l’analisi dei pattern spazio-temporali delle comunità richiede un elevato sforzo di campionamento. L’approccio molecolare risulta inoltre particolarmente utile per gli studi sulla dispersione larvale così come per monitoraggi finalizzati a garantire un sistema di fast alert sulla presenza di specie aliene invasive (NIS).